Aerosol and surface stability of HCoV-19 (SARS-CoV-2) compared to SARS-CoV-1
zu Deutsch: Aerosol und Oberflächen Überlebensdauer von HCoV-19 (SARS-CoV-2) verglichen mit SARS-CoV-1
Auftraggeber dieser Studie war das US Amerikanische Centers for Disease Control and Prevention unter Führung des Department of Health and Human Services
|
Veröffentlichung |
2020-03-13, in Version 2 |
|
MedRx |
https://www.medrxiv.org/content/10.1101/2020.03.09.20033217v2 |
|
DOI |
|
|
download PDF (5 Seiten) |
https://www.medrxiv.org/content/10.1101/2020.03.09.20033217v2.full.pdf |
|
The New England Journal of Medicine |
|
|
DOI |
|
|
download PDF (10 Seiten) |
https://www.nejm.org/doi/suppl/10.1056/NEJMc2004973/suppl_file/nejmc2004973_appendix.pdf |
Autorenliste
Neeltje van Doremalen, Trenton Bushmaker, Dylan H. Morris, Myndi G. Holbrook, Amandine Gamble, Brandi N. Williamson, Azaibi Tamin, Jennifer L. Harcourt, Natalie J. Thornburg, Susan I. Gerber, James O. Lloyd-Smith, Emmie de Wit, Vincent J. Munster
Inhalt
1 Inhaltsverzeichnis
2 Material und Methoden Seite 1-5
3 Ergänzende Tabelle 1 Seite 5
4 Ergänzende Abbildung 1 und 2 Seite 6
5 Ergänzende Abbildung 3 und 4 Seite 7
6 Ergänzende Abbildung 5 Seite 8
7 Ergänzende Hinweise Seite 8
8 Code- und Datenverfügbarkeit Seite 9
9 Danksagungen Seite 9
10
11
12
13
114 Ergänzende Methoden
15 Laborversuche
16 Viren und Titration
17 HCoV-19 nCoV-WA1-2020 (MN985325.1) (Holshue et al., 2020) und SARS-CoV-1 Tor2
18 (AY274119.3) (Marra et al., 2003) waren die in unserem Vergleich verwendeten Stämme. Lebensfähiges Virus in allen Oberflächen- und
19 Aerosolproben wurden durch Endpunkt-Titration an Vero E6-Zellen, wie zuvor beschrieben, quantifiziert (van
20 Doremalen et al., 2013).
21 Virusstabilität in Aerosolen
22 Die Virusstabilität in Aerosolen wurde wie zuvor beschrieben bei 65% relativer Luftfeuchtigkeit (RH) bestimmt und
23 21-23°C (Fischer et al., 2016). Kurz gesagt, Aerosole (<5 μm), die HCoV-19 (10 5,25 TCID 50 /mL) oder
24 SARS-CoV-1 (10 6,75-7 TCID50/mL) wurden mit einem 3-Strahl-Collison-Vernebler erzeugt und in einen Goldberg
25 Trommel zur Schaffung einer aerosolierten Umgebung. Die Aerosole wurden in der Goldberg-Trommel und den Proben
26 wurden bei 0, 30, 60, 120 und 180 Minuten nach der Aerosolisierung auf einem 47-mm-Gelatinefilter (Sartorius) gesammelt.
27 Filter wurden in 10 mL DMEM mit 10% FBS gelöst. Drei Wiederholungsexperimente wurden durchgeführt
28 durchgeführt.
29 Virusstabilität auf Oberflächen
30 Die Oberflächenstabilität wurde auf Kunststoff (Polypropylen, ePlastics), rostfreiem Stahl der Legierung AISI 304
31 (Metallrückstände), Kupfer (99,9%) (Metallrückstände) und Pappe (lokaler Anbieter) stellvertretend für eine Sorte
32 von Haushalts- und Krankenhaussituationen und wurde wie zuvor beschrieben bei 40% RH und 21-23°C durchgeführt
33 unter Verwendung eines Inokulums von 10 5 TCID 50 /mL (van Doremalen et al., 2013). Dieses Inokulum führte zu einem Zyklus-
34 Schwellenwerte (Ct) zwischen 20 und 22, die denen ähnlich sind, die bei Proben von menschlichen oberen und unteren
35 der Atemwege (Zou et al., 2020). Kurz gesagt, 50 μl des Virus wurden auf der Oberfläche abgelagert und bei
36 vordefinierte Zeitpunkte durch Hinzufügen von 1 mL DMEM. Die Stabilität auf Karton wurde durch Ablagerung von 50
37 μl des Virus auf der Oberfläche und die Rückgewinnung des Inokulums durch Abstriche von der Oberfläche, der Abstrich wurde
38 1 ml DMEM. Für jede Oberfläche wurden drei Wiederholungsexperimente durchgeführt.
239 Statistische Analysen
40 Beschreibung des Bayes'schen Regressionsmodells
41 Die Dauer der Nachweisbarkeit hängt erwartungsgemäß vom Ausgangsinokulum und der Probenahmemethode ab. An
42 die inhärente Stabilität der Viren bewerten, schätzten wir die Zerfallsraten der lebensfähigen Virustiter unter Verwendung eines
43 Bayes'sches Regressionsmodell. Dieser Modellierungsansatz ermöglichte es uns, Unterschiede in der anfänglichen
44 Inokulumniveaus über Replikate hinweg, sowie Intervallzensierung von Titerdaten und anderen Quellen von
45 experimentelles Rauschen. Das Modell liefert Schätzungen der posterioren Verteilungen der viralen Zerfallsraten und Halbwertszeiten
46 in den verschiedenen Versuchsbedingungen - d.h. Schätzungen der Bandbreite der plausiblen Werte für diese
47 Parameter angesichts unserer Daten, mit einer Schätzung der Gesamtunsicherheit (Gelman et al., 2013).
48 In der folgenden Modellnotation bedeutet das Symbol ~, dass eine Zufallsvariable verteilt ist gemäß
49 für die gegebene Verteilung. Normalverteilungen werden als Normal (Mittelwert, Standardabweichung) parametrisiert.
50 Positiv-beschränkte Normalverteilungen ("Halbnormal") werden als Halbnormal(modus) parametrisiert,
51 Standardabweichung). Wir verwenden <Distribution Name>CDF(x, Parameter), um die kumulative Verteilung zu bezeichnen
52 Funktion einer Wahrscheinlichkeitsverteilung, also z.B. NormalCDF(5, 0, 1) ist der Wert des Normalen(0, 1)
53 kumulative Verteilungsfunktion bei 5.
54 Unsere Daten bestehen aus 10 experimentellen Bedingungen: 2 Viren (HCoV-19 und SARS-CoV-1) bei 5
55 Umweltbedingungen (Aerosole, Kunststoff, Edelstahl, Kupfer und Pappe). Jede hat drei Replikate,
56 und mehrere Zeitpunkte für jede Wiederholung. Wir analysieren die beiden Viren getrennt. Für jedes bezeichnen wir durch
57 y ijk der gemessene log 10-Titer im experimentellen Zustand i während der Replikation j zum Zeitpunkt k. Zur Konstruktion unserer
58 Wahrscheinlichkeitsfunktion, wir müssen die Wahrscheinlichkeit kennen, mit der eine gegebene log 10-Titer-Messung y ijk gegeben
59 Werte der Parameter.
60 Da unsere Titerdaten in Inkrementen von 1/n Vertiefungen log 10 TCID 50 /mL geschätzt und aufgezeichnet werden, wobei n Vertiefungen
61 ist die Anzahl der Vertiefungen, die für die Endpunkt-Titration verwendet werden, unsere log 10-Titerwerte sind intervallzensiert - nur bekannt
62 bis innerhalb eines Bereichs von Breite 1/n Wells . Darüber hinaus gibt es ein gewisses Messrauschen beim Titrationsprozess
63 selbst.
64 Um dies zu modellieren, nehmen wir an, dass in jeder Versuchsbedingung i ein echter zugrunde liegender log 10-Titer
65 x ijk, der mit einem gewissen Maß an experimentellem Rauschen oder Fehler gemessen wird ε ijk und dann als Intervall beobachtet wird-
66 zensierter Wert y ijk ≈ x ijk + ε ijk . Wir modellieren die Messfehler ε ijk als normalverteilt mit einem Standard
67 Abweichung σ i die von allen Proben unter den gegebenen Versuchsbedingungen geteilt wird, spiegelt die Tatsache wider, dass einige
68 Versuchsaufbauten können mehr oder weniger laut sein als andere.
369 ε ijk ~ Normal(0, σ i )
70 Wir modellieren die Wahrscheinlichkeit der Beobachtung eines intervallzensierten log 10-Titerwertes y ijk bei einem wahren Basiswert
71
log 10 titer x ijk und eine Messfehler-Standardabweichung σ i as:
72 P(y ijk | x ijk , σ i ) = NormalCDF(y ijk , x ijk , σ i ) - NormalCDF(y ijk - 1/n Vertiefungen , x ijk , σ i )
73 Dies spiegelt die Wahrscheinlichkeit bei einem wahren Wert x ijk plus den Messfehler x ijk + ε ijk liegt zwischen
74 y ijk - 1/n Brunnen und y ijk . Aufgrund des verwendeten log 10-Titer-Imputationsverfahrens ist ein Titer in diesem Bereich am wahrscheinlichsten
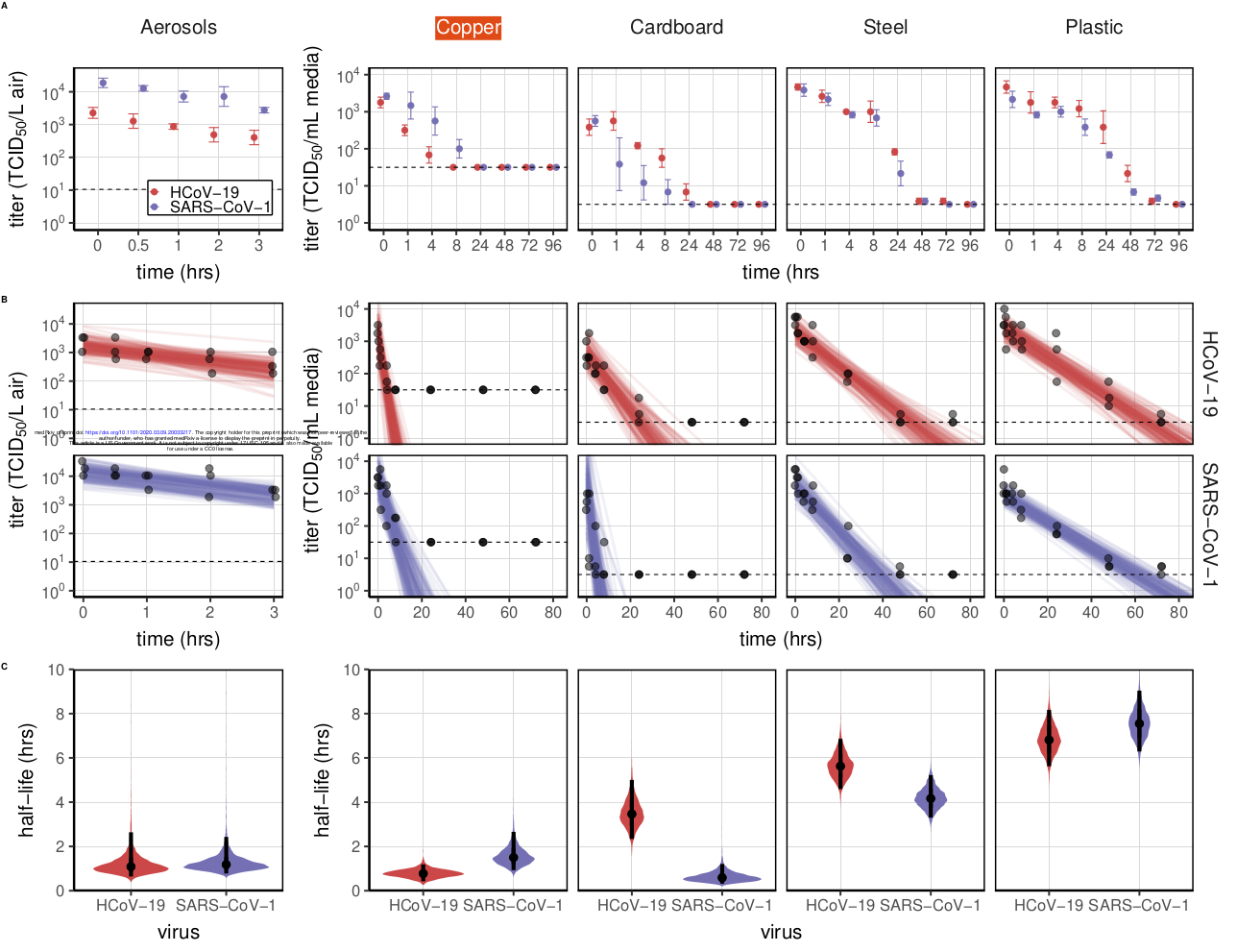
75 werden aufgerundet und als y ijk angegeben.
76
77
Die Nachweisgrenze unseres Experiments liegt bei 0,5 log 10 TCID 50 /mL. Die Wahrscheinlichkeit der Beobachtung eines
nicht nachweisbarer gemessener log 10-Titerwert y ijk bei einem wahren log 10-Titerwert x ijk ist gegeben durch:
78 P(y ijk ≤ 0,5 | x ijk , σ i ) = NormalCDF(0,5, x ijk , σ i )
79 Wir modellieren dann jede Wiederholung j für die experimentelle Bedingung i so, dass sie mit einem echten anfänglichen log 10-Titer beginnt
80 x ij (0) = x ij0 . Wir nehmen an, dass Viren im experimentellen Zustand i exponentiell mit einer Rate λ i über die Zeit t zerfallen.
81 Daraus folgt, dass
x ij (t) = x ij0 - λ i t
82
83
84
wobei t k der k-te gemessene Zeitpunkt ist.
Modellieren Sie frühere Verteilungen
85 Wir platzieren eine schwach informative Normale Vorverteilung auf dem anfänglichen log 10 Titer x ij0, um auszuschließen
86 unplausibel große oder kleine Werte (z.B. in diesem Fall nicht nachweisbare log 10-Titer oder log 10-Titer, die viel höher sind als
87 der hinterlegten Konzentration), wobei die Daten Schätzungen innerhalb plausibler Bereiche ermöglichen:
88
89
90
x ij0 ~ Normal(4,5, 2,5)
Wir legten ebenfalls einen schwach informativen Halbnormalvorrang auf die exponentiellen Zerfallsraten λ i :
λ i ~ Halbnormal(0.5, 4)
4Wir haben den Standardabweichungen des Experiments ein schwach informatives Halbnormal vorangestellt.
91
92
Fehlerverteilungen σ i :
σ i ~ Halbnormal(0, 2)
93
94
Markov-Kette Monte-Carlo-Methoden
95 Wir zogen posteriore Proben mit Stan, der einen No-U-Turn-Sampler (eine Form von Markov
96 Kette Monte Carlo). Wir führten vier Replikationsketten aus zufälligen Anfangsbedingungen für 2000 Iterationen durch, mit
97 die ersten 1000 Iterationen als Aufwärm-/Anpassungsphase. Wir speicherten die letzten 1000 Iterationen von jeder Kette,
98 mit insgesamt 4000 posterioren Proben. Wir beurteilten die Konvergenz durch die Inspektion von Spurenparzellen und
99 Untersuchung von R̂ und Statistiken zur effektiven Stichprobengröße (n eff ) (R̂ für alle Parameter ≤ 1,003, n eff für alle Parameter
100 ≥28% der gesamten Stichproben).
101 Ergänzende Tabelle und Abbildungen
102 Tabelle 1. Nachträgliche Medianschätzungen und 95% glaubwürdige Intervalle (2,5%-97,5% Quantilsbereich) für Halbwertszeiten
103 von HCoV-19 und SARS-CoV-1 in Aerosolen und auf verschiedenen Oberflächen, sowie eine mittlere Schätzung und 95%
104 glaubwürdiges Intervall für die Differenz zwischen den beiden Halbwertszeiten (HCoV-19 - SARS-CoV-1).
HCoV-19 SARS-CoV-1 HCoV-19 - SARS-CoV-1
Halbwertszeit (Std.) Halbwertszeit (Std.) Differenz (Std.)
Material Medianwert 2,5% 97,5% Medianwert 2,5% 97,5% Medianwert 2,5% 97,5%
Aerosole 1,09 0,64 2,64 1,18 0,778 2,43 -0,0913 -1,35 1,39
Kupfer 0,774 0,427 1,19 1,5 0,929 2,66 -0,735 -1,91 -0,0339
Pappe 3,46 2,34 5 0,587 0,317 1,21 2,85 1,58 4,41
Stahl 5,63 4,59 6,86 4,16 3,3 5,22 1,46 0,00127 2,96
Kunststoff 6,81 5,62 8,17 7,55 6,29 9,04 -0,722 -2,64 1,16
105
106 Die Abbildungen S1-S5 (unten) zeigen Bayes'sche Übereinstimmungen mit individuellen Replikat-Virus-Zerfallsdaten für jedes Virus. Repliziert
107 werden in Panel-Spalten angezeigt, Viren in Panel-Zeilen. Zeilen sind 50 zufällige Ziehungen pro Tafel aus dem hinteren
5108 Verteilung der angepassten Linien, um den Grad der Unsicherheit zu zeigen. Die Zeitachse ist bis zur letzten Zeit, die bis
109 erreichen unter den betrachteten Versuchsbedingungen einen nicht nachweisbaren Titer.
110
111 Abbildung S1. Individuelle Wiederholungspassungen für Aerosole. Spalten zeigen Replikate, Zeilen zeigen Virus (HCoV-19
112 oben, SARS-CoV-1 unten). Die Linien sind 50 zufällige Ziehungen pro Feld aus der hinteren Verteilung der angepassten
113 Zeilen, um den Grad der Unsicherheit zu zeigen.
114
115 Abbildung S2. Individuelle Wiederholungspassungen für Kunststoff. Spalten zeigen Replikate, Zeilen zeigen Virus (HCoV-19 oben),
116 SARS-CoV-1 unten). Bei den Linien handelt es sich um 50 zufällige Ziehungen pro Feld aus der hinteren Verteilung der eingepassten Linien,
117, um den Grad der Unsicherheit aufzuzeigen.
6118
119 Abbildung S3. Individuelle Wiederholungspassungen für Stahl. Spalten zeigen Replikate, Zeilen zeigen Virus (HCoV-19 oben,
120 SARS-CoV-1 unten). Bei den Linien handelt es sich um 50 zufällige Ziehungen pro Feld aus der hinteren Verteilung der eingepassten Linien,
121, um den Grad der Unsicherheit aufzuzeigen.
122
123 Abbildung S4. Individuelle Wiederholungspassungen für Kupfer. Die Spalten zeigen Replikate, die Zeilen zeigen Virus (HCoV-19 oben),
124 SARS-CoV-1 unten). Bei den Linien handelt es sich um 50 zufällige Ziehungen pro Feld aus der hinteren Verteilung der eingepassten Linien,
125, um den Grad der Unsicherheit aufzuzeigen. Fits sind für SARS-CoV-1 wesentlich schlechter als für HCoV-19, und die Daten
126 nicht einem linearen Abwärtstrend über die Zeit folgen, was darauf hindeutet, dass der Unterschied in den beobachteten Zerfallsraten
127 mit Vorsicht zu interpretieren.
7128
129 Abbildung S5. Individuelle Wiederholungspassungen für Karton. Spalten zeigen Replikate, Zeilen zeigen Virus (HCoV-19
130 oben, SARS-CoV-1 unten). Die Linien sind 50 zufällige Ziehungen pro Feld aus der hinteren Verteilung der angepassten
131 Zeilen, um den Grad der Unsicherheit aufzuzeigen. Die Passungen sind für SARS-CoV-1 wesentlich schlechter als für HCoV-19, und
132 Daten folgen nicht einem linearen Abwärtstrend über die Zeit, was darauf hindeutet, dass der Unterschied in den beobachteten Zerfallsraten
133 ist mit Vorsicht zu interpretieren.
134 Ergänzende Hinweise
135 Fischer, R.J., Bushmaker, T., Judson, S., Munster, V.J., 2016. Vergleich der Aerosol-Stabilität von 2
136 Stämme des Zaire-Ebolavirus von den Ausbrüchen 1976 und 2013. J. Infizieren. Dis. 214, 290–293.
137
138
139
Gelman, A., Carlin, J.B., Stern, H.S., Dunson, D.B., Vehtari, A., Rubin, D.B., 2013. Bayessche Daten
Analyse, dritte Ausgabe. CRC Presse.
Holshue, M.L., DeBolt, C., Lindquist, S., Lofy, K.H., Wiesman, J., Bruce, H., Spitters, C., Ericson, K.,
140 Wilkerson, S., Tural, A., Diaz, G., Cohn, A., Fox, L., Patel, A., Gerber, S.I., Kim, L., Tong, S., Lu,
141 X., Lindstrom, S., Pallansch, M.A., Weldon, W.C., Biggs, H.M., Uyeki, T.M., Pillai, S.K., 2020.
142 Erster Fall des 2019 neuartigen Coronavirus in den Vereinigten Staaten. N. Engl. J. Med. 382, 929–936.
143 Marra, M.A., Jones, S.J.M., Astell, C.R., Holt, R.A., Brooks-Wilson, A., Butterfield, Y.S.N., Khattra, J.,
144 Asano, J.K., Barber, S.A., Chan, S.Y., Cloutier, A., Coughlin, S.M., Freeman, D., Girn, N., Griffith,
145 O.L., Leach, S.R., Mayo, M., McDonald, H., Montgomery, S.B., Pandoh, P.K., Petrescu, A.S,
146 Robertson, A.G., Schein, J.E., Siddiqui, A., Smailus, D.E., Stott, J.M., Yang, G.S., Plummer, F.,
147 Andonov, A., Artsob, H., Bastien, N., Bernard, K., Booth, T.F., Bowness, D., Czub, M., Drebot, M.,
148 Fernando, L., Flick, R., Garbutt, M., Gray, M., Grolla, A., Jones, S., Feldmann, H., Meyers, A., Czub, M., Czub, M., Drebot, M,
8149 Kabani, A., Li, Y., Normand, S., Stroher, U., Tipples, G.A., Tyler, S., Vogrig, R., Ward, D., Watson,
150 B., Brunham, R.C., Krajden, M., Petric, M., Skowronski, D.M., Upton, C., Roper, R.L., 2003. Die
151 Genomsequenz des SARS-assoziierten Coronavirus. Wissenschaft 300, 1399-1404.
152 van Doremalen, N., Buschmacher, T., Münster, V., 2013. Stabilität des respiratorischen Syndroms im Nahen Osten
153 Coronavirus (MERS-CoV) unter verschiedenen Umweltbedingungen. Eurosurveillance 18, 20590.
154 Zou, L., Ruan, F., Huang, M., Liang, L., Huang, H., Hong, Z., Yu, J., Kang, M., Song, Y., Xia, J., Guo,
155 Q., Song, T., He, J., Yen, H.-L., Peiris, M., Wu, J., 2020. SARS-CoV-2 Viruslast im oberen
156 Respirationsproben von infizierten Patienten. N. Engl. J. Med. Im Druck.
157
158
Verfügbarkeit von Codes und Daten
Code und Daten zur Reproduktion der Bayes'schen Schätzungsergebnisse und zur Erzeugung entsprechender Zahlen sind
159 online archiviert bei OSF: <Link einfügen> und auf Github verfügbar: <Link einfügen>
160 Danksagungen
161 Wir möchten Kwe Claude Yinde und Michael Letko für ihre experimentelle Unterstützung danken. Diese
162 Forschungsarbeiten wurden durch das Intramural Research Program des National Institute of Allergy unterstützt und
163 Infektionskrankheiten (NIAID), Nationale Gesundheitsinstitute (NIH). JOL-S und AG wurden unterstützt von der
164 Defense Advanced Research Projects Agency DARPA PREEMPT # D18AC00031, und JOL-S war
165 unterstützt von der U.S. National Science Foundation (DEB-1557022) und der Strategic Environmental
166 Forschungs- und Entwicklungsprogramm (SERDP, RC-2635) des US-Verteidigungsministeriums. Die Ergebnisse
167 und die Schlussfolgerungen in diesem Bericht sind die des/der Verfasser(s) und stellen nicht unbedingt die offizielle Position dar
168 der Centers for Disease Control and Prevention. Namen von bestimmten Verkäufern, Herstellern oder Produkten
169 sind für die öffentliche Gesundheit und zu Informationszwecken aufgenommen worden; die Aufnahme bedeutet nicht die Billigung der
170 Verkäufer, Hersteller oder Produkte von den Centers for Disease Control and Prevention oder den USA
171 Ministerium für Gesundheit und Humandienste